Einleitung
Dieses Dokument soll einem Anwender des Packages
rOstluft::rOstluft einen Überblick der enthaltenen
Funktionalität bieten und die gängigsten Arbeitsabläufe aufzeigen.
Der Fokus dieses Packages liegt in der Bereitstellung von Daten aus verschiedenen Datenquellen in einem einheitlichen Format zur Analyse der Luftqualität. Ausserdem enthält es Werkzeuge für einige übliche Aufgaben, die während solchen Analysen anfallen. Diese sind Umrechnungen zwischen verschiedenen Mittelungsintervallen, Statistische Methoden mit Berücksichtung der Datenverfügbarkeit, Umwandlung von Volumen- und Massenkonzentrationen und lesen von Daten vorliegend in verschiedenen Formaten.
Installation
Der Quellcode von rOstluft ist auf github gehosted. Die einfachste Variante ist die Installation mit Hilfe des Packages devtools:
#install.packages("devtools")
devtools::install_github("Ostluft/rOstluft")Zusätzlich muss das Package aws.s3 manuell aus dem
cloudyr Repositorium installiert werden, weil die CRANR Version veraltet
ist:
install.packages("aws.s3", repos = c("cloudyr" = "http://cloudyr.github.io/drat"))Ist dies wegen Einschränkungen durch Firewalls oder Proxies nicht möglich. Muss der Quellcode manuell von github heruntergeladen werden (Clone or download > Download as ZIP), entpackt und manuell installiert werden. Allerdings bestehen Abhängigkeiten zu Packages die auf CRAN bereitgestellt werden. Können auch keine CRAN Packages installiert werden, müssen zuerst alle CRAN Abhängkigkeiten und deren Abhängigkeiten installiert werden.
Zusätzlich besteht noch die Github Abhängkigkeit zu rOstluft.data. Dieses Packages muss auf die gleiche Weise zuerst installiert werden mit folgenden Schritten:
download.file("https://github.com/Ostluft/rOstluft/archive/master.zip", "rOstluft-master.zip")
download.file("https://github.com/Ostluft/rOstluft.data/archive/master.zip", "rOstluft.data-master.zip")
install.packages("devtools")
install.packages("aws.s3", repos = c("cloudyr" = "http://cloudyr.github.io/drat"))
deps <- c('dplyr', 'tidyr', 'lubridate', 'R6', 'rappdirs', 'tibble', 'base64url', 'forcats',
'fs', 'purrr', 'readr', 'stringr', 'stringi', 'sp', 'rgdal', 'rlang', 'magrittr')
for (p in deps) {
install.packages(p)
}
devtools::install_local("rOstluft.data-master.zip", dependencies = FALSE)
devtools::install_local("rOstluft-master.zip", dependencies = FALSE)Falls das installieren von rOstluft scheitert, fehlt vermutlich eine Abhängigkeit. Welche das ist, kann der Fehlermeldung entnommen werden.
Nach der Installation kann das Packages verwendet werden:
Abfrage von Daten
Ostluft Amazon AWS S3
Die zentrale Datenablage innerhalb von Ostluft erfolgt auf Amazon AWS S3.Aus Lizenztechnischen Gründen kann das Bucket nicht öffentlich zugänglich gemacht werden. Die Zugangsdaten werden von Jörg Sintermann vergeben. Die Zugangsdaten werden am einfachsten über eine .Renvirion Datei im Verzeichnis des RStudio Projektes oder im HOME Verzeichnis des Users1 dem Package zugänglich gemacht. Inhalt der .Renvirion Datei:
AWS_ACCESS_KEY_ID = "XXXXXXXXXXXXXXXXXXXX"
AWS_SECRET_ACCESS_KEY = "XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX"
AWS_DEFAULT_REGION = "eu-central-1"Weitere Möglichkeiten sind in der Dokumentation von aws.signature zu finden.
Sämtliche Daten die einmal von Amazon S3 geöffnet wurden, werden lokal auf dem Rechner gespeichert. Bei jedem folgenden Zugriff wird nur überprüft, ob die Daten noch identisch sind.
Der Zugriff auf die Daten erfügt dann über ein store Objekt, welches auf folgende Art initialisiert wird:
store <- storage_s3_rds("tutorial", format = format_rolf(), bucket = "rostluft", prefix = "aqmet")
#> store tutorial initialized under 'C:\Users\tom\AppData\Local/rOstluft/tutorial'
# oder nutze den vordefinierten store (name ist optional, default ist "aqmet")
store <- store_aqmet("tutorial")Dieses store Objekt verfügt über verschiedene Methoden, zur Abfrage von Daten sind jedoch nur zwei von Bedeutung:
-
$get_content(): holt eine Übersicht über alle im store enthaltene Daten -
$get(): Aktuelle Datenabfrage. Welche Daten geholt werden, muss über Funktionsargumente definiert werden
content <- store$get_content()
content
#> # A tibble: 220,692 × 6
#> year interval site parameter unit n
#> <dbl> <fct> <fct> <fct> <fct> <int>
#> 1 2019 y1 8400_0040 NO2 µg/m3 1
#> 2 2022 y1 9658_0020 NH3 µg/m3 1
#> 3 2022 y1 8880_0030 NH3 µg/m3 1
#> 4 2022 y1 8535_0010 NH3 µg/m3 1
#> 5 2022 y1 WeF_Weid NH3 µg/m3 1
#> 6 2022 y1 Tan_NABEL NH3 µg/m3 1
#> 7 2022 y1 StE_Rödelbach NO2 µg/m3 1
#> 8 2022 y1 StE_Rödelbach NH3 µg/m3 1
#> 9 2022 y1 SNs_Wellrüti NH3 µg/m3 1
#> 10 2022 y1 Hae_Buo NH3 µg/m3 1
#> # ℹ 220,682 more rows
dplyr::sample_n(content, 10)
#> # A tibble: 10 × 6
#> year interval site parameter unit n
#> <dbl> <fct> <fct> <fct> <fct> <int>
#> 1 1997 y1 Ziz_Neulöser T_min_d1 °C 1
#> 2 2009 y1 Dübendorf-Empa T_max_h1 °C 1
#> 3 2021 d1 Säntis Hr_nb_min10 1 363
#> 4 2004 y1 Gra_Marktplatz WVs_max_min30 m/s 1
#> 5 1999 d1 Ott_Waldstation T_max_min30 °C 365
#> 6 2013 d1 Zch_Rosengartenstrasse O3_max_h1 µg/m3 89
#> 7 1995 y1 Zürich/Kloten T_min_min10 °C 1
#> 8 2018 y1 Hor_Sekundarschule Hr %Hr 1
#> 9 2019 m1 Rap_Tüchelweier RainDur_nb_min30 1 12
#> 10 2004 y1 StG_Rorschacher Strasse SO2_min_min30 µg/m3 1Eine Datenabfrage holt immer die Daten von einem komplettem Jahr einer Station für einen bestimmten Mittelungszeitraum:
store$get(site = "Zch_Schimmelstrasse", year = 2014, interval = "min30")
#> # A tibble: 261,315 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2014-01-01 00:00:00 Zch_Schimmelstrasse CO min30 ppm 0.637
#> 2 2014-01-01 00:00:00 Zch_Schimmelstrasse Hr min30 %Hr 87.4
#> 3 2014-01-01 00:00:00 Zch_Schimmelstrasse NO2 min30 ppb 28.1
#> 4 2014-01-01 00:00:00 Zch_Schimmelstrasse NO min30 ppb 61.5
#> 5 2014-01-01 00:00:00 Zch_Schimmelstrasse NOx min30 ppb 89.6
#> 6 2014-01-01 00:00:00 Zch_Schimmelstrasse O3 min30 ppb 0.798
#> 7 2014-01-01 00:00:00 Zch_Schimmelstrasse PM10 min30 µg/m3 102.
#> 8 2014-01-01 00:00:00 Zch_Schimmelstrasse PN min30 1/cm3 25971
#> 9 2014-01-01 00:00:00 Zch_Schimmelstrasse RainDur min30 min 0
#> 10 2014-01-01 00:00:00 Zch_Schimmelstrasse SO2 min30 ppb 2.12
#> # ℹ 261,305 more rowsJedes der Argumente kann auch ein Vector sein mit mehreren Werte. Es wird dann Kombination aller Möglichkeiten abgefragt.
sites <- c("Zch_Schimmelstrasse", "Zch_Rosengartenstrasse", "Zch_Stampfenbachstrasse")
data <- store$get(site = sites, year = 2014:2015, interval = "min30")
data
#> # A tibble: 1,391,156 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2014-01-01 00:00:00 Zch_Rosengartenstrasse Hr min30 %Hr 92.7
#> 2 2014-01-01 00:00:00 Zch_Rosengartenstrasse NO2 min30 ppb 18.6
#> 3 2014-01-01 00:00:00 Zch_Rosengartenstrasse NO min30 ppb 42.8
#> 4 2014-01-01 00:00:00 Zch_Rosengartenstrasse NOx min30 ppb 61.4
#> 5 2014-01-01 00:00:00 Zch_Rosengartenstrasse O3 min30 ppb 0.776
#> 6 2014-01-01 00:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 298.
#> 7 2014-01-01 00:00:00 Zch_Rosengartenstrasse RainDur min30 min 0
#> 8 2014-01-01 00:00:00 Zch_Rosengartenstrasse T min30 °C -2.18
#> 9 2014-01-01 00:00:00 Zch_Rosengartenstrasse p min30 hPa 966.
#> 10 2014-01-01 00:00:00 Zch_Rosengartenstrasse O3 min30 µg/m3 1.55
#> # ℹ 1,391,146 more rows
dplyr::sample_n(data, 10)
#> # A tibble: 10 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2015-09-23 11:30:00 Zch_Rosengartenstrasse Hr min30 %Hr 76.2
#> 2 2015-07-10 03:00:00 Zch_Schimmelstrasse p min30 hPa 975.
#> 3 2014-08-21 14:00:00 Zch_Rosengartenstrasse NOx min30 ppb 72.3
#> 4 2015-05-15 21:00:00 Zch_Rosengartenstrasse Hr min30 %Hr 90.0
#> 5 2014-07-17 00:00:00 Zch_Rosengartenstrasse T min30 °C 20
#> 6 2015-03-02 01:00:00 Zch_Schimmelstrasse SO2 min30 ppb 0.570
#> 7 2014-01-05 11:00:00 Zch_Schimmelstrasse SO2 min30 ppb 0.762
#> 8 2015-02-07 22:30:00 Zch_Schimmelstrasse Hr min30 %Hr 68.2
#> 9 2015-10-16 09:30:00 Zch_Stampfenbachstrasse NOx min30 ppb 41.7
#> 10 2014-10-07 23:00:00 Zch_Stampfenbachstrasse p min30 hPa 965.Die letzte Funktionalität von $get() ist die Filterung
der Daten vor der Rückgabe. Mit dem Argument “filter” als
dplyr::filter() kompatibler Ausdruck:
store$get(site = sites, year = 2014, interval = "min30", filter = parameter == "PM10")
#> # A tibble: 51,319 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2014-01-01 00:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 298.
#> 2 2014-01-01 00:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 557.
#> 3 2014-01-01 01:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 308.
#> 4 2014-01-01 01:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 169.
#> 5 2014-01-01 02:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 128.
#> 6 2014-01-01 02:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 82.9
#> 7 2014-01-01 03:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 38.3
#> 8 2014-01-01 03:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 32.6
#> 9 2014-01-01 04:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 32.4
#> 10 2014-01-01 04:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 31.7
#> # ℹ 51,309 more rowsLokales Arbeiten mit den AWS S3 Daten
Ein Nachteil des S3 Storage ist, dass bei jeder Datenabfrage eine Internetverbindung zur Überprüfung, ob aktualisierte Daten verfügbar sind, vorhanden sein muss. Selbst wenn die Daten bereits lokal gespeichert sind. Nicht nur wird eine Internetverbindung benötigt, die Überprüfung braucht auch eine kurze Zeit. Hat man alle notwendigen Daten bereits herunter geladen, kann man jedoch mit einem lokalen Store arbeiten. Praktischerweise hat der S3 Store eine Funktion, welche einen lokalen Store zurückgibt.
lokal <- store$get_local_storage()Dieser verfügt über die gleichen Funktionalität wie der S3 Store:
lokal$get_content()
#> # A tibble: 220,692 × 6
#> year interval site parameter unit n
#> <dbl> <fct> <fct> <fct> <fct> <int>
#> 1 2019 y1 8400_0040 NO2 µg/m3 1
#> 2 2022 y1 9658_0020 NH3 µg/m3 1
#> 3 2022 y1 8880_0030 NH3 µg/m3 1
#> 4 2022 y1 8535_0010 NH3 µg/m3 1
#> 5 2022 y1 WeF_Weid NH3 µg/m3 1
#> 6 2022 y1 Tan_NABEL NH3 µg/m3 1
#> 7 2022 y1 StE_Rödelbach NO2 µg/m3 1
#> 8 2022 y1 StE_Rödelbach NH3 µg/m3 1
#> 9 2022 y1 SNs_Wellrüti NH3 µg/m3 1
#> 10 2022 y1 Hae_Buo NH3 µg/m3 1
#> # ℹ 220,682 more rows
lokal$get(site = sites, year = 2014, interval = "min30", filter = parameter == "PM10")
#> # A tibble: 51,319 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2014-01-01 00:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 298.
#> 2 2014-01-01 00:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 557.
#> 3 2014-01-01 01:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 308.
#> 4 2014-01-01 01:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 169.
#> 5 2014-01-01 02:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 128.
#> 6 2014-01-01 02:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 82.9
#> 7 2014-01-01 03:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 38.3
#> 8 2014-01-01 03:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 32.6
#> 9 2014-01-01 04:00:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 32.4
#> 10 2014-01-01 04:30:00 Zch_Rosengartenstrasse PM10 min30 µg/m3 31.7
#> # ℹ 51,309 more rowsDem aufmerksamen Leser ist vermutlich aufgefallen, dass die
Inhaltsübersicht von $get_content() nicht dem lokalen
Inhalt entspricht. Es ist immer noch die Übersicht welche Daten in S3
verfügbar wären. Will man wissen welche Daten lokal verfügbar sind,
hilft einem die Funktion $list_chunks() weiter.
tibble::glimpse(lokal$list_chunks())
#> Rows: 6
#> Columns: 7
#> $ chunk_name <fs::path> "min30/bWluMzDCu1pjaF9Sb3NlbmdhcnRlbnN0cm…
#> $ interval <chr> "min30", "min30", "min30", "min30", "min30", "…
#> $ site <chr> "Zch_Rosengartenstrasse", "Zch_Rosengartenstra…
#> $ year <chr> "2014", "2015", "2014", "2015", "2014", "2015"
#> $ local.path <fs::path> "C:/Users/tom/AppData/Local/rOstluft/tutorial/…
#> $ local.modification_time <dttm> 2023-12-25 16:14:33, 2023-12-25 16:14:33,…
#> $ local.size <fs::bytes> 808.46K, 924.81K, 1.22M, 1.22M, 1.22M, 1.13M…Auch der S3 Store verfügt über die $list_chunks()
Funktion.
tibble::glimpse(store$list_chunks())
#> Rows: 16,070
#> Columns: 11
#> $ chunk_name <fs::path> "d1/ZDHCu01hdV9Qw7xudMK7MTk5MA", "d1/ZDHC…
#> $ interval <chr> "d1", "d1", "d1", "d1", "d1", "d1", "d1", "d1"…
#> $ site <chr> "Mau_Pünt", "Mau_Pünt", "Mau_Pünt", "Mau_Pünt"…
#> $ year <chr> "1990", "1991", "1992", "1993", "1994", "1995"…
#> $ s3.key <chr> "aqmet/data/d1/ZDHCu01hdV9Qw7xudMK7MTk5MA.rds"…
#> $ s3.lastmodified <chr> "2023-04-26T18:15:28.000Z", "2023-04-26T18:15:…
#> $ s3.etag <chr> "348548bdf0411ed6c385654884752ef9", "4e887d5d0…
#> $ s3.size <fs::bytes> 15.71K, 27.03K, 29.17K, 28.24K, 16.88K, …
#> $ local.path <fs::path> NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, N…
#> $ local.modification_time <dttm> NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, N…
#> $ local.size <fs::bytes> NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, …Im normal Fall ist Nutzung von $get_content() über
$list_chunks() zu bevorzugen. Die Rückgabe von
$get_content() enthält die gemessen Parameter und die
Anzahl der gültigen Punkte. Bei $list_chunks() hingegen
sind einige interne Informationen über den Store enthalten.
Um die Vorbereitung um mit einem lokalen Store zu arbeiten zu
vereinfachen verfügt der S3 Store über die Funktion
$download(...). Die dot Argumente werden als Filter auf die
Rückgabe von $list_chunks() angewendet. Ohne Argumente wird
der komplette Store heruntergeladen. Folgendes Beispiel lädt samtliche
30 Minutenmittelwerte für die Station Rosengartenstrasse nach dem Jahr
2015 (Achtung == verwenden, es sind Filter Ausdrücke!):
store$download(site == "Zch_Rosengartenstrasse", interval == "min30", year > 2015)
#> list()
lokal$list_chunks() %>% dplyr::select("site", "year", "interval")
#> # A tibble: 13 × 3
#> site year interval
#> <chr> <chr> <chr>
#> 1 Zch_Rosengartenstrasse 2020 min30
#> 2 Zch_Rosengartenstrasse 2021 min30
#> 3 Zch_Rosengartenstrasse 2022 min30
#> 4 Zch_Rosengartenstrasse 2017 min30
#> 5 Zch_Rosengartenstrasse 2018 min30
#> 6 Zch_Rosengartenstrasse 2019 min30
#> 7 Zch_Rosengartenstrasse 2014 min30
#> 8 Zch_Rosengartenstrasse 2015 min30
#> 9 Zch_Rosengartenstrasse 2016 min30
#> 10 Zch_Stampfenbachstrasse 2014 min30
#> 11 Zch_Stampfenbachstrasse 2015 min30
#> 12 Zch_Schimmelstrasse 2014 min30
#> 13 Zch_Schimmelstrasse 2015 min30Eigene Daten in einem lokalen Store
Der aqmet S3 Store enthält nur bereinigte Daten von abgeschlossenen Jahren. Werden Daten benötigt, die nicht in S3 vorhanden sind, bietet sich die Nutzung eines seperaten lokalen Stores an:
my_store = storage_local_rds("eigene_daten", format_rolf(), read.only = FALSE)
#> Local store eigene_daten initialized under 'C:\Users\tom\AppData\Local/rOstluft/eigene_daten'In diesen kann man nun Daten mit der Store Funktion
$put() oder der Hilfsfunktion
import_directory() importieren:
examples_path <- system.file("extdata", package = "rOstluft.data")
import_directory(my_store, examples_path, read_airmo_csv, glob = "*Jan.csv")
#> Importing 'C:/Users/tom/AppData/Local/R/cache/R/renv/library/rOstluft-c971cee0/R-4.3/x86_64-w64-mingw32/rOstluft.data/extdata/Zch_Stampfenbachstrasse_d1_2013_Jan.csv' with size 25.4K. File 1 of 3
#> Read 'C:/Users/tom/AppData/Local/R/cache/R/renv/library/rOstluft-c971cee0/R-4.3/x86_64-w64-mingw32/rOstluft.data/extdata/Zch_Stampfenbachstrasse_d1_2013_Jan.csv' in 0.61 seconds. Got 2170 data points
#> First put to storage. Save columns types to C:/Users/tom/AppData/Local/rOstluft/eigene_daten/columns.rds
#> Put data into store eigene_daten in 0.09 seconds
#> Importing 'C:/Users/tom/AppData/Local/R/cache/R/renv/library/rOstluft-c971cee0/R-4.3/x86_64-w64-mingw32/rOstluft.data/extdata/Zch_Stampfenbachstrasse_h1_2013_Jan.csv' with size 142K. File 2 of 3
#> Read 'C:/Users/tom/AppData/Local/R/cache/R/renv/library/rOstluft-c971cee0/R-4.3/x86_64-w64-mingw32/rOstluft.data/extdata/Zch_Stampfenbachstrasse_h1_2013_Jan.csv' in 0.08 seconds. Got 14116 data points
#> Put data into store eigene_daten in 0.13 seconds
#> Importing 'C:/Users/tom/AppData/Local/R/cache/R/renv/library/rOstluft-c971cee0/R-4.3/x86_64-w64-mingw32/rOstluft.data/extdata/Zch_Stampfenbachstrasse_min30_2013_Jan.csv' with size 258K. File 3 of 3
#> Read 'C:/Users/tom/AppData/Local/R/cache/R/renv/library/rOstluft-c971cee0/R-4.3/x86_64-w64-mingw32/rOstluft.data/extdata/Zch_Stampfenbachstrasse_min30_2013_Jan.csv' in 0.08 seconds. Got 28234 data points
#> Put data into store eigene_daten in 0.10 seconds
#> Finished import after 1.10 seconds
fn <- fs::path(examples_path, "Zch_Rosengartenstrasse_2010-2014.csv")
data <- read_airmo_csv(fn)
my_store$put(data)
#> # A tibble: 18 × 6
#> year interval site parameter unit n
#> <dbl> <fct> <fct> <fct> <fct> <int>
#> 1 2013 min30 Zch_Rosengartenstrasse NO µg/m3 4269
#> 2 2013 min30 Zch_Rosengartenstrasse NO2 µg/m3 4269
#> 3 2013 min30 Zch_Rosengartenstrasse NOx ppb 4269
#> 4 2013 min30 Zch_Rosengartenstrasse O3 µg/m3 4277
#> 5 2013 min30 Zch_Rosengartenstrasse PM10 µg/m3 4025
#> 6 2013 min30 Zch_Rosengartenstrasse Hr %Hr 4010
#> 7 2013 min30 Zch_Rosengartenstrasse p hPa 4291
#> 8 2013 min30 Zch_Rosengartenstrasse RainDur min 4291
#> 9 2013 min30 Zch_Rosengartenstrasse T °C 4010
#> 10 2014 min30 Zch_Rosengartenstrasse NO µg/m3 17415
#> 11 2014 min30 Zch_Rosengartenstrasse NO2 µg/m3 17415
#> 12 2014 min30 Zch_Rosengartenstrasse NOx ppb 17415
#> 13 2014 min30 Zch_Rosengartenstrasse O3 µg/m3 17366
#> 14 2014 min30 Zch_Rosengartenstrasse PM10 µg/m3 16788
#> 15 2014 min30 Zch_Rosengartenstrasse Hr %Hr 17517
#> 16 2014 min30 Zch_Rosengartenstrasse p hPa 17519
#> 17 2014 min30 Zch_Rosengartenstrasse RainDur min 17519
#> 18 2014 min30 Zch_Rosengartenstrasse T °C 17517Wie gewohnt kann erhält man mit $get_content() eine
Übersicht und die Daten mit $get():
my_store$get_content()
#> # A tibble: 126 × 6
#> year interval site parameter unit n
#> <dbl> <fct> <fct> <fct> <fct> <int>
#> 1 2014 min30 Zch_Rosengartenstrasse NO µg/m3 17415
#> 2 2014 min30 Zch_Rosengartenstrasse NO2 µg/m3 17415
#> 3 2014 min30 Zch_Rosengartenstrasse NOx ppb 17415
#> 4 2014 min30 Zch_Rosengartenstrasse O3 µg/m3 17366
#> 5 2014 min30 Zch_Rosengartenstrasse PM10 µg/m3 16788
#> 6 2014 min30 Zch_Rosengartenstrasse Hr %Hr 17517
#> 7 2014 min30 Zch_Rosengartenstrasse p hPa 17519
#> 8 2014 min30 Zch_Rosengartenstrasse RainDur min 17519
#> 9 2014 min30 Zch_Rosengartenstrasse T °C 17517
#> 10 2013 min30 Zch_Rosengartenstrasse NO µg/m3 4269
#> # ℹ 116 more rows
my_store$get(site = "Zch_Stampfenbachstrasse", interval = c("d1", "h1"), year = 2013) %>%
dplyr::arrange(.data$starttime, .data$parameter)
#> # A tibble: 16,286 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.381
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 ppm 0.327
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO h1 mg/m3 0.805
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO h1 ppm 0.692
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_max_min… d1 mg/m3 0.966
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_min_min… d1 mg/m3 0.190
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_nb_min30 d1 1 48
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr d1 %Hr 76.0
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr h1 %Hr 82.9
#> 10 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr_max_min… d1 %Hr 88.4
#> # ℹ 16,276 more rowsEinlesefunktionen von Daten
Um Daten in R einzulesen exisitieren für folgende Quellen bereits Funktionen:
- Ostluft:
read_airmo_csv()undread_airmo_dat()für Exporte aus der AIRMO - MeteoSchweiz:
read_smn()undread_smn_multiple()für SwissMetNet Exporte - ETHZ/IAC:
read_ethz_iac()Meteo Stationen auf dem Hönggerberg und dem Hauptgebäude CHN
Existiert für die vorliegenden Daten keine Funktion und man möchte die selber schreiben an dieser Stelle einige Hinweise und Tipps.
Verwende readr Funktionen und definiere die Zeichenkodierung
Ein generelles Problem beim einlesen von Textdateien ist die Zeichenkodierung.
Die meisten haben vermutlich “�” schon in Texten gesehen. Alle Daten im
aqmet Store und alle Rückgabewerte von read_xxx()
Funktionen sind UTF-8 kodiert. Mit dem readr Packages wird
bei korrekter Definition der Locale
der Text automatisch zu UTF-8 konvertiert. Im Gegensatz zu den R Base
Funktionen oderdata.table::fread().
Ausgabeformat rolf
Für ein reibungsloses Zusammenspiel mit den Stores zu garantieren muss das Format rolf eingehalten werden. rolf setzt einen tibble mit folgenden Spalten und Klassen voraus:
| Spalte | Klasse |
|---|---|
| “starttime” | POSIXct |
| “site” | factor |
| “parameter” | factor |
| “interval” | factor |
| “unit” | factor |
| “value” | double |
Selbst wenn in der Datei keine Einheiten enthalten sind, ist es
besser die Spalte unit mit NA zu initialisieren. Ein weiterer Trick ist
es die Spalten am Ende explizit zu selektionieren. Hier ein Beispiel,
wie es in read_smn() gelöst ist:
data <- dplyr::mutate(data,
stn = forcats::as_factor(.data$stn),
parameter = forcats::as_factor(.data$parameter),
interval = forcats::as_factor(interval),
unit = factor(NA)
)
dplyr::select(data, starttime = "time", site = "stn", "parameter", "interval", "unit", "value")Mit dem dplyr::select am Ende ist man sicher nur die
Spalten in der Rückgabe zu haben die man haben will und es ist einfach
noch Spalten umzubenennen und die Reihenfolge zu ändern.
Zusammenfügen von Daten im rolf Format
Die Verwendung von Factoren im rolf Format hat seine Vor- und
Nachteile. Der Hauptvorteil liegt bei schnelleren Lese- und
Schreiboperation, der grösste Nachteil beim Zusammenfügen der Daten.
Jedoch unterstützt seit dplyr v1.0.0 dplyr::bind_rows das
Zusammenfügen von Factor Spalten. Diese Funktion unterstützt ausserdem
Quasiquotation
von rlang2. Somit kann auch eine grosse Liste von
tibbles im rolf Format komfortable aneinander gefügt werden:
df1 <- read_airmo_csv(fs::path(examples_path, "Zch_Stampfenbachstrasse_h1_2013_Jan.csv"))
df2 <- read_airmo_csv(fs::path(examples_path, "Zch_Stampfenbachstrasse_d1_2013_Jan.csv"))
df_comb <- dplyr::bind_rows(df1, df2)
df_comb %>% dplyr::arrange(.data$starttime, .data$parameter)
#> # A tibble: 16,286 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO h1 mg/m3 0.805
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO h1 ppm 0.692
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.381
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 ppm 0.327
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr h1 %Hr 82.9
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr d1 %Hr 76.0
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO h1 µg/m3 80.5
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO h1 ppb 64.6
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO d1 µg/m3 17.6
#> 10 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO d1 ppb 14.1
#> # ℹ 16,276 more rows
# liste mit 20+ tibbles
df_list <- read_smn_multiple(fs::path(examples_path, "smn_multi.txt"), as_list = TRUE)
length(df_list)
#> [1] 28
df_comb <- dplyr::bind_rows(!!!df_list)
df_comb %>% dplyr::arrange(.data$starttime, .data$parameter)
#> # A tibble: 1,399 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2010-01-01 00:00:00 KLO dkl010b0 h1 NA 319
#> 2 2010-01-01 00:00:00 KLO fkl010b0 h1 NA 1.5
#> 3 2010-01-01 00:00:00 KLO gre000b0 h1 NA 0
#> 4 2010-01-01 00:00:00 KLO tre200b0 h1 NA 2.3
#> 5 2010-01-01 00:00:00 UEB dk1towb0 h1 NA 249
#> 6 2010-01-01 00:00:00 UEB fk1towb0 h1 NA 1.7
#> 7 2010-01-01 00:00:00 UEB ta1towb0 h1 NA 3.7
#> 8 2010-01-01 01:00:00 KLO dkl010b0 h1 NA 317
#> 9 2010-01-01 01:00:00 KLO fkl010b0 h1 NA 1.7
#> 10 2010-01-01 01:00:00 KLO gre000b0 h1 NA 0
#> # ℹ 1,389 more rowsdplyr::bind_rows() entfernt jedoch keine Duplikate aus
den Daten. Es hängt die Daten einfach aneinander. Möchte man Duplikate
entfernen, bzw. Daten aktualisieren mit neueren Daten muss man die
Funktion $merge() des Format Objekts benützen. Dieses wurde
beim Erzeugen des Store direkt initialisiert und ist als Feld
$format verfügbar. Alternativ kann ein seperates Format
Objekt erzeugt werden. Die Daten im ersten Datenframe werden über Daten
im zweiten Datenframe priorisiert.
# kopiere Daten und setze einige NA -> Resultat sollte NA enthalten
df1 <- df2
df1$value[1:150] <- NA
df_comb <- store$format$merge(df1, df2)
df_comb %>% dplyr::arrange(.data$starttime, .data$parameter)
#> # A tibble: 2,170 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 NA
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 ppm NA
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_max_min30 d1 mg/m3 NA
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_min_min30 d1 mg/m3 NA
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_nb_min30 d1 1 NA
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr d1 %Hr 76.0
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr_max_min30 d1 %Hr 88.4
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr_min_min30 d1 %Hr 50.3
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr_nb_min30 d1 1 48
#> 10 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO d1 µg/m3 17.6
#> # ℹ 2,160 more rows
rolf <- format_rolf()
df_comb <- rolf$merge(df1, df2)
df_comb %>% dplyr::arrange(.data$starttime, .data$parameter)
#> # A tibble: 2,170 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 NA
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 ppm NA
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_max_min30 d1 mg/m3 NA
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_min_min30 d1 mg/m3 NA
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO_nb_min30 d1 1 NA
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr d1 %Hr 76.0
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr_max_min30 d1 %Hr 88.4
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr_min_min30 d1 %Hr 50.3
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr_nb_min30 d1 1 48
#> 10 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO d1 µg/m3 17.6
#> # ℹ 2,160 more rowsDie $merge() Funktion unterstützt nur 2 Argumente. Muss
eine Liste von tibbles gemerget werden muss man
purrr::reduce() benützen. Mit Hilfe des Argument
.dir kann man die Priosierung setzen. Mit “forward”” haben
die Daten die zuerst in der Liste auftauchen Priorität, mit “backward”
die Letzten.
df_comb <- purrr::reduce(df_list, rolf$merge, .dir = "forward")
df_comb %>% dplyr::arrange(.data$starttime, .data$parameter)
#> # A tibble: 1,399 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2010-01-01 00:00:00 KLO dkl010b0 h1 NA 319
#> 2 2010-01-01 00:00:00 KLO fkl010b0 h1 NA 1.5
#> 3 2010-01-01 00:00:00 KLO gre000b0 h1 NA 0
#> 4 2010-01-01 00:00:00 KLO tre200b0 h1 NA 2.3
#> 5 2010-01-01 00:00:00 UEB dk1towb0 h1 NA 249
#> 6 2010-01-01 00:00:00 UEB fk1towb0 h1 NA 1.7
#> 7 2010-01-01 00:00:00 UEB ta1towb0 h1 NA 3.7
#> 8 2010-01-01 01:00:00 KLO dkl010b0 h1 NA 317
#> 9 2010-01-01 01:00:00 KLO fkl010b0 h1 NA 1.7
#> 10 2010-01-01 01:00:00 KLO gre000b0 h1 NA 0
#> # ℹ 1,389 more rowsMetadaten
Daten aus verschiedenen Quellen werden verschiedene Schemen für die Vergabe von Namen haben. MeteoSchweiz zum Beispiel kodiert im Parameter Namen gleichzeitig das Mittelungsinterval:
| Parameter | Interval | Messgrösse |
|---|---|---|
| ta1towb0 | Bürgerliche Stundenmittel | Lufttemperatur, Instrument 1 |
| ta1towh0 | Stundenmittel | Lufttemperatur, Instrument 1 |
| ta1tows0 | Momentanwerte | Lufttemperatur, Instrument 1 |
| fk1towz0 | Zehnminutenmittel | Windgeschwindigkeit skalar, Instrument 1 |
Um die Bezeichnungen zu normalisieren kommt es zu einem Zusammenspiel
von im Store bereitgestellte Daten und der Funktion
meta_apply(). Das Store Objekt verfügt über die Funktion
$get_meta(), welche im AWS S3 bereitgestellte Metadaten
zurück gibt. Wird $get_meta() ohne Argument aufgerufen
erhält man eine Liste mit sämtlichen Metadaten im Store. Kennt man
bereits den Namen kann man direkt die entsprechenden Metadaten
holen:
meta <- store$get_meta()
names(meta)
#> [1] "all" "ecmwf"
#> [3] "ethz" "hysplit"
#> [5] "hysplit_cluster_trajectories" "meteoschweiz"
#> [7] "meteotest" "nabel"
#> [9] "ostluft" "seepolizei"
meteoschweiz <- store$get_meta("meteoschweiz")
tibble::glimpse(meteoschweiz)
#> List of 1
#> $ meteoschweiz: tibble [279 × 15] (S3: tbl_df/tbl/data.frame)
#> ..$ site_short : chr [1:279] "TAE" "TAE" "TAE" "TAE" ...
#> ..$ site : chr [1:279] "Aadorf/Tänikon" "Aadorf/Tänikon" "Aadorf/Tänikon" "Aadorf/Tänikon" ...
#> ..$ Länge : chr [1:279] "8°54'" "8°54'" "8°54'" "8°54'" ...
#> ..$ Breite : chr [1:279] "47°29'" "47°29'" "47°29'" "47°29'" ...
#> ..$ x : num [1:279] 2710517 2710517 2710517 2710517 2710517 ...
#> ..$ y : num [1:279] 1259824 1259824 1259824 1259824 1259824 ...
#> ..$ masl : num [1:279] 539 539 539 539 539 539 539 539 539 539 ...
#> ..$ parameter_original: chr [1:279] "tre200s0" "ure200s0" "prestas0" "prestas0" ...
#> ..$ source : chr [1:279] "MeteoSchweiz" "MeteoSchweiz" "MeteoSchweiz" "MeteoSchweiz" ...
#> ..$ unit : chr [1:279] "°C" "%" "hPa" "hPa" ...
#> ..$ Beschreibung : chr [1:279] "Lufttemperatur" "Relative" "Luftdruck" "Luftdruck" ...
#> ..$ timezone_original : chr [1:279] "UTC" "UTC" "UTC" "UTC" ...
#> ..$ site_long : logi [1:279] NA NA NA NA NA NA ...
#> ..$ interval : chr [1:279] "min10" "min10" "min10" "min10" ...
#> ..$ parameter : chr [1:279] "T" "Hr" "p" "p" ...Die meta_apply() benützt die Metadaten als “Lookup
Table”. Es wird folgende Operation ausgeführt:
meta_apply(data, meta, data_src, data_dest, meta_key, meta_val)
data$data_dest = meta[meta$meta_key == data$data_src]$meta_valWobei die jeweiligen Spalten jeweils als Argument übergeben werden. Die Funktion verfügt über verschiende Modi um mit fehlenden Einträgen umzugehen. Im folgenden Beispiel werden bei den MeteoSchweiz Daten die “unit” Spalte basierend auf den “parameter” Spalte ausgefüllt. Da die Parameter noch die MeteoSchweiz Bezeichnungen haben müssen wir als Meta Key die Spalte “parameter_original” und “unit” als Value verwenden. Es werden die Daten aus rOstluft.data verwendet. In diesen fehlt in den Metadaten der MeteoSchweiz Parameter rre150z0 (Niederschlag). Der Default Modus “strict” stoppt das Script:
meteoschweiz <- readRDS(fs::path(examples_path, "meta_smn.rds"))
data <- read_smn(fs::path(examples_path, "smn.txt"), na.rm = FALSE)
data <- dplyr::arrange(data, .data$starttime, .data$parameter)
data
#> # A tibble: 60 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2018-01-01 00:50:00 CHU dkl010z0 min10 NA 216
#> 2 2018-01-01 00:50:00 CHU fkl010z0 min10 NA 1.7
#> 3 2018-01-01 00:50:00 CHU fkl010z1 min10 NA 3
#> 4 2018-01-01 00:50:00 CHU gre000z0 min10 NA 5
#> 5 2018-01-01 00:50:00 CHU hto000s0 min10 NA NA
#> 6 2018-01-01 00:50:00 CHU prestas0 min10 NA 946.
#> 7 2018-01-01 00:50:00 CHU rre150z0 min10 NA 0
#> 8 2018-01-01 00:50:00 CHU sre000z0 min10 NA 0
#> 9 2018-01-01 00:50:00 CHU tre200s0 min10 NA 2.3
#> 10 2018-01-01 00:50:00 CHU ure200s0 min10 NA 82.7
#> # ℹ 50 more rows
tryCatch({
meta_apply(data, meteoschweiz, "parameter", "unit", "parameter_original", "unit")
}, error = function(e) {
sprintf(e$message)
})
#> [1] "apply meta data$unit = meta[meta$parameter_original == data$parameter]$unit:\n missing keys in meta$parameter_original: rre150z0"Im einfachsten Fall werden Zeilen mit fehlenden Einträge einfach gelöscht. Dies geschieht mit dem Modus “drop”:
df <- meta_apply(data, meteoschweiz, "parameter", "unit", "parameter_original", "unit", mode = "drop")
#> Warning in meta_apply(data, meteoschweiz, "parameter", "unit", "parameter_original", : apply meta data$unit = meta[meta$parameter_original == data$parameter]$unit:
#> missing keys in meta$parameter_original: rre150z0
#> dropping missing prameters
df
#> # A tibble: 54 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2018-01-01 00:50:00 CHU dkl010z0 min10 ° 216
#> 2 2018-01-01 00:50:00 CHU fkl010z0 min10 m/s 1.7
#> 3 2018-01-01 00:50:00 CHU fkl010z1 min10 m/s 3
#> 4 2018-01-01 00:50:00 CHU gre000z0 min10 W/m2 5
#> 5 2018-01-01 00:50:00 CHU hto000s0 min10 cm NA
#> 6 2018-01-01 00:50:00 CHU prestas0 min10 hPa 946.
#> 7 2018-01-01 00:50:00 CHU sre000z0 min10 min 0
#> 8 2018-01-01 00:50:00 CHU tre200s0 min10 °C 2.3
#> 9 2018-01-01 00:50:00 CHU ure200s0 min10 % 82.7
#> 10 2018-01-01 01:00:00 CHU dkl010z0 min10 ° 209
#> # ℹ 44 more rowsSollte der Parameter für spätere Bearbeitung behalten werden, kommt der Modus “keep” zum Zug:
df <- meta_apply(data, meteoschweiz, "parameter", "unit", "parameter_original", "unit", mode = "keep")
#> Warning in meta_apply(data, meteoschweiz, "parameter", "unit", "parameter_original", : apply meta data$unit = meta[meta$parameter_original == data$parameter]$unit:
#> missing keys in meta$parameter_original: rre150z0
#> keeping values: NA
df
#> # A tibble: 60 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2018-01-01 00:50:00 CHU dkl010z0 min10 ° 216
#> 2 2018-01-01 00:50:00 CHU fkl010z0 min10 m/s 1.7
#> 3 2018-01-01 00:50:00 CHU fkl010z1 min10 m/s 3
#> 4 2018-01-01 00:50:00 CHU gre000z0 min10 W/m2 5
#> 5 2018-01-01 00:50:00 CHU hto000s0 min10 cm NA
#> 6 2018-01-01 00:50:00 CHU prestas0 min10 hPa 946.
#> 7 2018-01-01 00:50:00 CHU rre150z0 min10 NA 0
#> 8 2018-01-01 00:50:00 CHU sre000z0 min10 min 0
#> 9 2018-01-01 00:50:00 CHU tre200s0 min10 °C 2.3
#> 10 2018-01-01 00:50:00 CHU ure200s0 min10 % 82.7
#> # ℹ 50 more rowsMit Hilfe des Modus “replace” kann eine zusätzliche “Lookup Table” übergeben werden. Werte in dieser Tabelle haben Priorität über Werte in der Meta Tabelle. Im Beispiel wird die fehlende Einheit für rre150z0 bereit gestellt und die Einheit für dkl010z0 von ° zu deg überschrieben.
df <- meta_apply(data, meteoschweiz, "parameter", "unit", "parameter_original", "unit",
mode = "replace", replacements = list(rre150z0 = "mm", dkl010z0 = "deg"))
#> apply meta data$unit = meta[meta$parameter_original == data$parameter]$unit:
#> missing keys in meta$parameter_original: rre150z0
#> replacements used: dkl010z0, rre150z0
df
#> # A tibble: 60 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2018-01-01 00:50:00 CHU dkl010z0 min10 deg 216
#> 2 2018-01-01 00:50:00 CHU fkl010z0 min10 m/s 1.7
#> 3 2018-01-01 00:50:00 CHU fkl010z1 min10 m/s 3
#> 4 2018-01-01 00:50:00 CHU gre000z0 min10 W/m2 5
#> 5 2018-01-01 00:50:00 CHU hto000s0 min10 cm NA
#> 6 2018-01-01 00:50:00 CHU prestas0 min10 hPa 946.
#> 7 2018-01-01 00:50:00 CHU rre150z0 min10 mm 0
#> 8 2018-01-01 00:50:00 CHU sre000z0 min10 min 0
#> 9 2018-01-01 00:50:00 CHU tre200s0 min10 °C 2.3
#> 10 2018-01-01 00:50:00 CHU ure200s0 min10 % 82.7
#> # ℹ 50 more rowsHier ein Beispiel für die komplette Überführung der MeteoSchweiz Daten, inkl. manuelle Umbennung von rre150z0 und Änderung der Einheit für die Windrichtung (WD) von ° zu deg:
df <- meta_apply(data, meteoschweiz, "parameter", "unit", "parameter_original", "unit",
mode = "replace", replacements = list(rre150z0 = "mm", dkl010z0 = "deg"))
#> apply meta data$unit = meta[meta$parameter_original == data$parameter]$unit:
#> missing keys in meta$parameter_original: rre150z0
#> replacements used: dkl010z0, rre150z0
df <- meta_apply(df, meteoschweiz, "parameter", "parameter", "parameter_original", "parameter",
mode = "replace", replacements = list(rre150z0 = "Niederschlag"))
#> apply meta data$parameter = meta[meta$parameter_original == data$parameter]$parameter:
#> missing keys in meta$parameter_original: rre150z0
#> replacements used: rre150z0
df <- meta_apply(df, meteoschweiz, "site", "site", "site_short", "site")
df
#> # A tibble: 60 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2018-01-01 00:50:00 Chur WD min10 deg 216
#> 2 2018-01-01 00:50:00 Chur WVs min10 m/s 1.7
#> 3 2018-01-01 00:50:00 Chur WVs_max min10 m/s 3
#> 4 2018-01-01 00:50:00 Chur StrGlo min10 W/m2 5
#> 5 2018-01-01 00:50:00 Chur SnowDep min10 cm NA
#> 6 2018-01-01 00:50:00 Chur p min10 hPa 946.
#> 7 2018-01-01 00:50:00 Chur Niederschlag min10 mm 0
#> 8 2018-01-01 00:50:00 Chur SunDur min10 min 0
#> 9 2018-01-01 00:50:00 Chur T min10 °C 2.3
#> 10 2018-01-01 00:50:00 Chur Hr min10 % 82.7
#> # ℹ 50 more rowsopenair kompatibles Wide Format
Das Long Format ist nicht für jede Analyse geeignet. Aus diesem Grund
enthält rOstluft die Funktionalität Daten im rolf Format in ein openair kompatibles
Wide Format umzuwandeln. Ein Nachteil am Wide Format ist, dass
Informationen die zu einer Spalte gehören, wie die Einheit einer
Messgrösse, verloren gehen. Dies führt auch dazu, das man nicht die
gleiche Messgrösse mit unterschiedlichen Einheiten in den Daten haben
kann. Ausser man nennt die Messgrösse um. Die Funktion
rolf_to_openair() geht folgende Kompromisse ein: Die
Einheiten werden entfernt, aber im Attribute “units” des Datenframes
gespeichert. Messgrössen mit der Einheit ppb oder ppm werden ebenfalls
entfernt (Dieser Automatismus lässt sich mit dem Argument keep_ppb
abschalten). Openair nimmt an, dass alle Messgrössen als
Massenkonzentrationen vorliegen. Weitere Konventionen in openair sind
das die Zeitinformation in der Spalte “date” als Date oder
POSIXct vorliegen, die Windgeschwindigkeit in der Spalte
“ws” und die Windrichtung in der Spalte “wd”. Sämtliche Character und
Factor Spalten dienen als Gruppierungsspalten. Diese werden von openair
ignoriert oder gar entfernt, wenn nicht das Argument type benützt wird.
Dieses erlaubt die explizite Nutzung einer Spalte als
Gruppierungskriterium. Es können mehrere Gruppierungskriterien getrennt
durch ein Komma definiert werden. Die openair Funktion
openair::cutData() übernimmt die Gruppierung.
data <- read_airmo_csv(fs::path(examples_path, "Zch_Stampfenbachstrasse_2010-2014.csv"))
wide <- rolf_to_openair(data)
tibble::glimpse(wide)
#> Rows: 87,626
#> Columns: 16
#> $ date <dttm> 2010-01-01 00:00:00, 2010-01-01 00:30:00, 2010-01-01 01:00:00…
#> $ site <fct> Zch_Stampfenbachstrasse, Zch_Stampfenbachstrasse, Zch_Stampfen…
#> $ CO <dbl> 0.513, 0.609, 0.822, 1.063, 0.861, 0.760, 0.633, 0.666, 0.780,…
#> $ NO <dbl> 9.50, 26.85, 59.95, 85.55, 72.04, 49.74, 26.50, 36.18, 60.38, …
#> $ NO2 <dbl> 37.0, 46.5, 54.6, 60.5, 58.8, 52.9, 49.0, 50.9, 56.3, 48.8, 47…
#> $ NOx <dbl> 26.9, 45.8, 76.6, 100.2, 88.5, 67.5, 46.9, 55.6, 77.8, 72.6, 5…
#> $ O3 <dbl> 11.812, 7.989, 2.111, 1.410, 2.517, 2.416, 3.606, 1.617, 2.600…
#> $ PM10 <dbl> 49.7, 82.3, 152.9, 169.2, 190.1, 158.9, 93.0, 54.9, 58.7, 81.7…
#> $ SO2 <dbl> 2.36, 3.13, 4.19, 4.60, 3.97, 2.71, 2.29, 2.46, 2.86, 3.36, 4.…
#> $ Hr <dbl> 87.9, 88.6, 91.1, 91.8, 92.0, 92.4, 92.8, 92.0, 90.4, 91.5, 92…
#> $ p <dbl> 942, 942, 942, 942, 942, 942, 941, 941, 941, 941, 940, 940, 94…
#> $ RainDur <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ StrGlo <dbl> 7.49e-03, 7.27e-03, 1.02e-02, 7.81e-03, 1.22e-02, 1.57e-02, 1.…
#> $ T <dbl> 5.61, 5.57, 5.35, 5.39, 5.11, 4.81, 4.58, 4.80, 5.38, 5.12, 4.…
#> $ wd <dbl> 159.57, 147.91, 258.24, 2.90, 27.75, 35.41, 40.24, 164.05, 182…
#> $ ws <dbl> 0.904, 0.314, 0.630, 0.625, 1.905, 2.281, 2.245, 0.846, 0.361,…Diese Daten können nun direkt in openair verwendet werden:
openair::windRose(wide)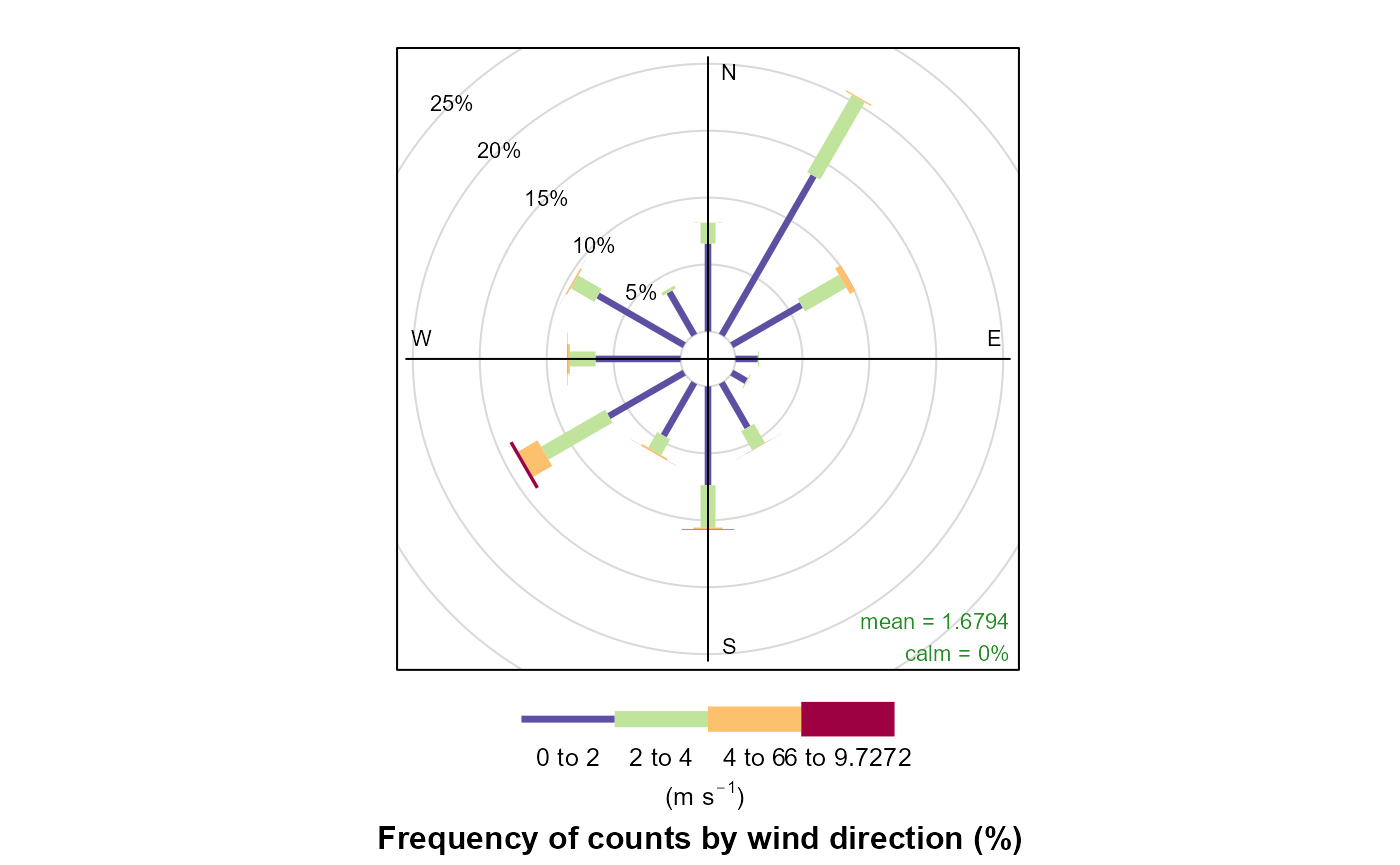
openair::pollutionRose(wide, "NO2", type="year")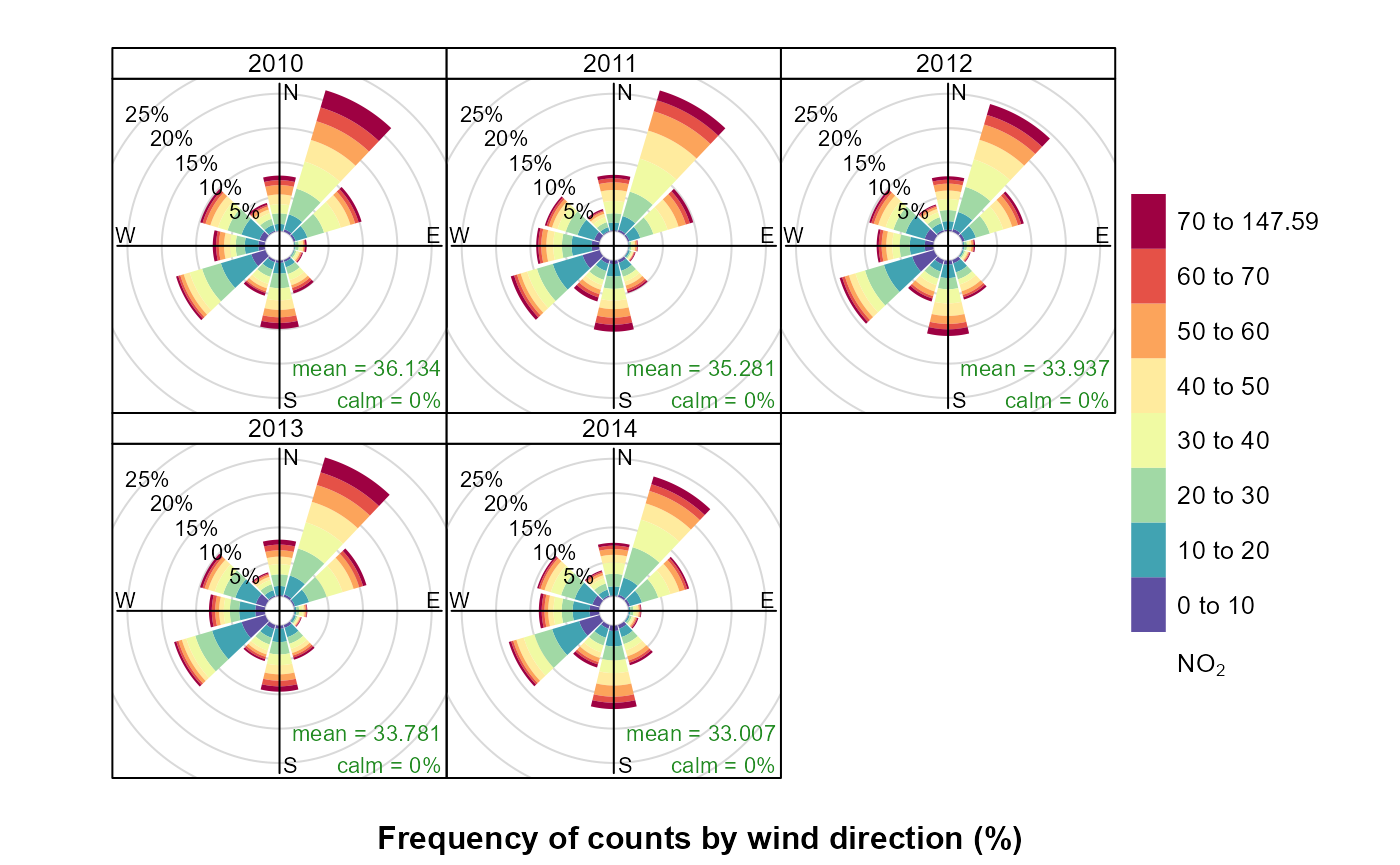
openair_to_rolf() konventiert Daten zurück ins rolf
Format. Der Nutzer muss die fehlenden Informationen bereitstellen:
openair_to_rolf(wide, interval = "min30", units = attr(wide, "units"))
#> # A tibble: 1,226,764 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2010-01-01 00:00:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.513
#> 2 2010-01-01 00:30:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.609
#> 3 2010-01-01 01:00:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.822
#> 4 2010-01-01 01:30:00 Zch_Stampfenbachstrasse CO min30 mg/m3 1.06
#> 5 2010-01-01 02:00:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.861
#> 6 2010-01-01 02:30:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.760
#> 7 2010-01-01 03:00:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.633
#> 8 2010-01-01 03:30:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.666
#> 9 2010-01-01 04:00:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.780
#> 10 2010-01-01 04:30:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.654
#> # ℹ 1,226,754 more rowsrolf_to_openair_single() bietet die Möglichkeit einen
bestimmten Parameter rauszupicken:
rolf_to_openair_single(data, "NO2", unit = "µg/m3", keep_interval = TRUE)
#> # A tibble: 86,883 × 5
#> date site interval unit NO2
#> <dttm> <fct> <fct> <fct> <dbl>
#> 1 2010-01-01 00:00:00 Zch_Stampfenbachstrasse min30 µg/m3 37.0
#> 2 2010-01-01 00:30:00 Zch_Stampfenbachstrasse min30 µg/m3 46.5
#> 3 2010-01-01 01:00:00 Zch_Stampfenbachstrasse min30 µg/m3 54.6
#> 4 2010-01-01 01:30:00 Zch_Stampfenbachstrasse min30 µg/m3 60.5
#> 5 2010-01-01 02:00:00 Zch_Stampfenbachstrasse min30 µg/m3 58.8
#> 6 2010-01-01 02:30:00 Zch_Stampfenbachstrasse min30 µg/m3 52.9
#> 7 2010-01-01 03:00:00 Zch_Stampfenbachstrasse min30 µg/m3 49.0
#> 8 2010-01-01 03:30:00 Zch_Stampfenbachstrasse min30 µg/m3 50.9
#> 9 2010-01-01 04:00:00 Zch_Stampfenbachstrasse min30 µg/m3 56.3
#> 10 2010-01-01 04:30:00 Zch_Stampfenbachstrasse min30 µg/m3 48.8
#> # ℹ 86,873 more rowsDie weitere Optionen für die Funktion rolf_to_openair()
sind in der Dokumentation zu finden.
Berechnung von Statistiken
Statistiken und die Verdichtung von Daten ist ein grundlegender
Bestandteil einer Datenanalyse. In rOstluft gibt es die Funktion
resample() zur Berechnung von Statistiken von einem
Interval zu einem anderen. Die Funktion
calculate_statstable() erlaubt die Definition von
Berechnungen über mehrere verschiedene Intervale, ist aber weniger
flexibel.
resample
resample() aggregiert die Daten in folgenden
Schritten:
- Aufsplitten der Daten in Serien (site, parameter, interval, unit)
-
pad_serie(): Auffüllen von Lücken mit NA (kann übersprungen werden) - Gruppiere Serie mit neuem Interval mit Hilfe von
lubridate::floor_date() - Berechne definierte Statistiken für den Parameter
-
- kombiniere die neu berechneten Serien
data <- read_airmo_csv(fs::path(examples_path, "Zch_Stampfenbachstrasse_min30_2013_Jan.csv"))
# Behalte nur Massenkonzentrationen
data <- dplyr::filter(data, !(.data$unit == "ppb" | .data$unit == "ppm"))
statistics <- list(
"default_statistic" = "drop",
"O3" = list("mean", "perc95", "n", "min", "max"),
"RainDur" = "sum",
"WD" = "wind.direction",
"WVs" = "wind.speed_scalar",
"WVv" = "wind.speed_vector"
)
# Monatsmittelwerte
resample(data, statistics, "m1")
#> # A tibble: 9 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 m1 µg/m3 2.12e+1
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_95%_min… m1 µg/m3 6.44e+1
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_min30 m1 1 1.49e+3
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_min_min… m1 µg/m3 1.04e+0
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_min… m1 µg/m3 7.67e+1
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse RainDur m1 min 6.26e+3
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVs m1 m/s 1.69e+0
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVv m1 m/s 4.57e-1
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WD m1 ° 2.58e+2
# Tagesmittelwerte
resample(data, statistics, "d1") %>%
dplyr::arrange(.data$starttime)
#> # A tibble: 279 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 d1 µg/m3 42.9
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_95%_min… d1 µg/m3 70.3
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_min30 d1 1 48
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_min_min… d1 µg/m3 2.58
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_min… d1 µg/m3 74.1
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse RainDur d1 min 495.
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVs d1 m/s 2.22
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVv d1 m/s 1.98
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WD d1 ° 227.
#> 10 2013-01-02 00:00:00 Zch_Stampfenbachstrasse O3 d1 µg/m3 36.0
#> # ℹ 269 more rowsresample() verfügt über die Funktionalität
Datenverfügbarkeit und Lücken mit den Argumenten
data_thresh und max_gap zu
berücksichtigen:
# Jahresmittelwerte nur mit Daten vom Januar
resample(data, statistics, "y1")
#> # A tibble: 9 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 y1 µg/m3 2.12e+1
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_95%_min… y1 µg/m3 6.44e+1
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_min30 y1 1 1.49e+3
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_min_min… y1 µg/m3 1.04e+0
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_min… y1 µg/m3 7.67e+1
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse RainDur y1 min 6.26e+3
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVs y1 m/s 1.69e+0
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVv y1 m/s 4.57e-1
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WD y1 ° 2.58e+2
# Jahresmittelwerte 80% Datenverfügbarkeit => all NA
resample(data, statistics, "y1", data_thresh = 0.8)
#> # A tibble: 9 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 y1 µg/m3 NA
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_95%_min30 y1 µg/m3 NA
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_min30 y1 1 NA
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_min_min30 y1 µg/m3 NA
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_min30 y1 µg/m3 NA
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse RainDur y1 min NA
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVs y1 m/s NA
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVv y1 m/s NA
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WD y1 ° NA
# Jahresmittelwerte 10 Tage Lücke = 48' 30min Werte => all NA
resample(data, statistics, "y1", max_gap = 480)
#> # A tibble: 9 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 y1 µg/m3 NA
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_95%_min30 y1 µg/m3 NA
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_min30 y1 1 NA
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_min_min30 y1 µg/m3 NA
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_min30 y1 µg/m3 NA
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse RainDur y1 min NA
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVs y1 m/s NA
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WVv y1 m/s NA
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse WD y1 ° NA
default_statistic
In der Liste werden für jeden Parameter die Statistiken definiert,
welche angewendet werden sollen. Der Listeneintrag
default_statistic wird auf alle nicht eingetragenen
Parameter angewendet. Die Statistik drop schliesst einen Parameter aus
den Resultaten aus. Dies erlaubt die schnelle Berechnung von Statistiken
für ein paar wenige Parametern. Gleichzeitig ist es auch möglich die
gleichen Statistiken auf viele Parameter anzuwenden und ein paar wenige
Ausnahmen zu definieren:
statistics <- list(
"default_statistic" = "mean",
"RainDur" = "sum",
"StrGlo" = "max",
"WD" = "drop",
"WVv" = "drop"
)
resample(data, statistics, "m1")
#> # A tibble: 11 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO m1 mg/m3 4.36e-1
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse Hr m1 %Hr 7.97e+1
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO m1 µg/m3 2.41e+1
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO2 m1 µg/m3 4.04e+1
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 m1 µg/m3 2.12e+1
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse p m1 hPa 9.64e+2
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse PM10 m1 µg/m3 2.63e+1
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse RainDur m1 min 6.26e+3
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse SO2 m1 µg/m3 2.54e+0
#> 10 2013-01-01 00:00:00 Zch_Stampfenbachstrasse StrGlo_ma… m1 W/m2 5.12e+2
#> 11 2013-01-01 00:00:00 Zch_Stampfenbachstrasse T m1 °C 2.12e+0Automatismen und Einschränkungen
Die Funktion benennt für die meisten Statistiken den Parameter gemäss dem AIRMO Schema3 um. Die Statistiken mean, median, sum, sd, percentile benennen den Paremeter nicht um und sind somit nicht kombinierbar.
Das Auffüllen der Lücken kann mittels des Argument
skip_padding übersprungen werden. Zusätzlich kann mittels
start_date, end_date und
drop_last das Füllen kontrolliert werden. Normalerweise
nimmt die Funktion das letzte Datum und füllt bis zum letzten neuen
Intervall auf. Werden zum Beispiel Tagesmittelwerte berechnet und es
sind nur Daten bis zum 22. Dezember 2017 10:00 Uhr vorhanden, wird der
letzte Wert in der Serie 2017-12-22 00:00 sein. Wird eine komplette
Jahresreihe benötigt, kann entweder das end_date auf
2017-12-31 oder auf 2018-01-01 und drop_last auf TRUE
gesetzt werden.
Windmittelung
Ein weiterer Spezialfall sind die Windberechnungen. Für die vektorielle Mittelung müssen die Paremeter zusammengefasst werden. Es müssen für alle drei Statistiken (wind.direction, wind.speed_vector, wind.speed_scalar) die Parameter definiert werden. Ist nur eine der Geschwindigkeiten vorhanden, wird die andere auf Basis der Vorhandenen berechnet. Zusätzlich ist die Windmittelung nicht kombinierbar mit anderen Statistiken.
Statstable
Werden viele verschiedene und mehrstufige Statistiken benötigt ist
die Berechnung mit resample() umständlich. Die Funktion
calculate_statstable() hingegen bietet diese
Funktionalität, hat aber folgende Einschränkungen:
- Datenreihen werden immer auf komplette Jahre aufgefüllt
- max_gap wird nur auf Berechnungen zu Jahresintervallen berücksichtigt und in Tagen definiert
- data_thresh ist für alle Berechnungen identisch.
- Intervall h8gl erwartet Intervall h1 als Basis
- Die Verwendung von
default_statisticund_input_führt schnell zu unerwarteten Ergebnissen
Zur Analyse von Luftqualität sind diese Einschränkungen kein Problem,
bzw. sogar erwünscht. Die Funktion calculate_statstable()
erwartet wie der Name schon vermuten lässt die Defintion der Statistiken
in einer Tabellenform. Die Tabelle hat die vier Spalten “parameter”,
“statistic”, “from”, “to”. Jede Zeile enthält eine zu berechnende
Statistik für einen Parameter vom Basis Interval (“from”) zum neuen
Interval (“to”). Die Tabelle wird dann gruppiert zuerst mit “from”, dann
mit “to” und zuletzt mit Parameter. Auf diese Weise wird eine Liste
generiert die mit resample() kompatibel ist. Ist keine
default_statistic definiert wird automatisch
default_statistic = "drop" zur Liste hinzugefügt. In
welcher Reihenfolge die verschiedenen “from” Intervalle berechnet
werden, kann optional mit Hilfe des Argument order definiert werden. Der
Default Wert ist
c("input", "h1", "h8gl", "d1", "m1", "y1"). Um die Tabelle
in einer kompakten Form zu erstellen kann in jeder Zelle mehrere Werte
getrennt durch “,” zusammengefasst werden. Hier die Tabelle zur
Berechnung der LRV Grenzwerte aus 30min Werten (entspricht 6 Aufrufe von
resample()):
lrv_table <- tibble::tribble(
~parameter, ~statistic, ~from, ~to,
"SO2, NO2, PM10", "mean", "input", "y1",
"SO2, NO2", "perc95", "input", "y1",
"O3", "perc98", "input", "m1",
"O3", "mean", "input", "h1",
"O3", "n>120", "h1", "y1",
"SO2, NO2, CO, PM10", "mean", "input", "d1",
"SO2", "n>100", "d1", "y1",
"NO2", "n>80", "d1", "y1",
"CO", "n>8", "d1", "y1",
"PM10", "n>50", "d1", "y1"
)
knitr::kable(lrv_table)| parameter | statistic | from | to |
|---|---|---|---|
| SO2, NO2, PM10 | mean | input | y1 |
| SO2, NO2 | perc95 | input | y1 |
| O3 | perc98 | input | m1 |
| O3 | mean | input | h1 |
| O3 | n>120 | h1 | y1 |
| SO2, NO2, CO, PM10 | mean | input | d1 |
| SO2 | n>100 | d1 | y1 |
| NO2 | n>80 | d1 | y1 |
| CO | n>8 | d1 | y1 |
| PM10 | n>50 | d1 | y1 |
Wird diese Tabelle verwendet resultiert eine Liste mit h1, m1 und y1 Einträgen, welche die berechneten Grössen enthält:
data <- read_airmo_csv(fs::path(examples_path, "Zch_Stampfenbachstrasse_min30_2017.csv"))
# Berechne Massenkonzentrationen aus Volumenkonzentrationen
data <- calculate_mass_concentrations(data)
stats <- calculate_statstable(data, lrv_table)
stats
#> $y1
#> # A tibble: 10 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2017-01-01 00:00:00 Zch_Stampfenbachstrasse NO2 y1 µg/m3 30.4
#> 2 2017-01-01 00:00:00 Zch_Stampfenbachstrasse NO2_95%_min… y1 µg/m3 68.8
#> 3 2017-01-01 00:00:00 Zch_Stampfenbachstrasse PM10 y1 µg/m3 15.9
#> 4 2017-01-01 00:00:00 Zch_Stampfenbachstrasse SO2 y1 µg/m3 1.05
#> 5 2017-01-01 00:00:00 Zch_Stampfenbachstrasse SO2_95%_min… y1 µg/m3 2.17
#> 6 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_h1>120 y1 1 81
#> 7 2017-01-01 00:00:00 Zch_Stampfenbachstrasse CO_nb_d1>8 y1 1 0
#> 8 2017-01-01 00:00:00 Zch_Stampfenbachstrasse NO2_nb_d1>80 y1 1 1
#> 9 2017-01-01 00:00:00 Zch_Stampfenbachstrasse PM10_nb_d1>… y1 1 8
#> 10 2017-01-01 00:00:00 Zch_Stampfenbachstrasse SO2_nb_d1>1… y1 1 0
#>
#> $m1
#> # A tibble: 12 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 62.5
#> 2 2017-02-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 79.3
#> 3 2017-03-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 94.4
#> 4 2017-04-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 105.
#> 5 2017-05-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 128.
#> 6 2017-06-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 134.
#> 7 2017-07-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 113.
#> 8 2017-08-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 109.
#> 9 2017-09-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 82.6
#> 10 2017-10-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 74.9
#> 11 2017-11-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 67.6
#> 12 2017-12-01 00:00:00 Zch_Stampfenbachstrasse O3_98%_min30 m1 µg/m3 71.8
#>
#> $h1
#> # A tibble: 8,760 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 1.45
#> 2 2017-01-01 01:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 1.35
#> 3 2017-01-01 02:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 1.02
#> 4 2017-01-01 03:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 1.54
#> 5 2017-01-01 04:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 2.41
#> 6 2017-01-01 05:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 2.14
#> 7 2017-01-01 06:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 2.41
#> 8 2017-01-01 07:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 2.67
#> 9 2017-01-01 08:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 3.74
#> 10 2017-01-01 09:00:00 Zch_Stampfenbachstrasse O3 h1 µg/m3 6.52
#> # ℹ 8,750 more rows
#>
#> $d1
#> # A tibble: 1,460 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2017-01-01 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.381
#> 2 2017-01-02 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.339
#> 3 2017-01-03 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.349
#> 4 2017-01-04 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.248
#> 5 2017-01-05 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.278
#> 6 2017-01-06 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.282
#> 7 2017-01-07 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.378
#> 8 2017-01-08 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.349
#> 9 2017-01-09 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.388
#> 10 2017-01-10 00:00:00 Zch_Stampfenbachstrasse CO d1 mg/m3 0.314
#> # ℹ 1,450 more rowsNur die m1 und y1 sind für die LRV relevant:
lrv <- dplyr::bind_rows(stats$y1, stats$m1)| starttime | site | parameter | interval | unit | value |
|---|---|---|---|---|---|
| 2017-01-01 | Zch_Stampfenbachstrasse | NO2 | y1 | µg/m3 | 30 |
| 2017-01-01 | Zch_Stampfenbachstrasse | NO2_95%_min30 | y1 | µg/m3 | 69 |
| 2017-01-01 | Zch_Stampfenbachstrasse | PM10 | y1 | µg/m3 | 16 |
| 2017-01-01 | Zch_Stampfenbachstrasse | SO2 | y1 | µg/m3 | 1 |
| 2017-01-01 | Zch_Stampfenbachstrasse | SO2_95%_min30 | y1 | µg/m3 | 2 |
| 2017-01-01 | Zch_Stampfenbachstrasse | O3_nb_h1>120 | y1 | 1 | 81 |
| 2017-01-01 | Zch_Stampfenbachstrasse | CO_nb_d1>8 | y1 | 1 | 0 |
| 2017-01-01 | Zch_Stampfenbachstrasse | NO2_nb_d1>80 | y1 | 1 | 1 |
| 2017-01-01 | Zch_Stampfenbachstrasse | PM10_nb_d1>50 | y1 | 1 | 8 |
| 2017-01-01 | Zch_Stampfenbachstrasse | SO2_nb_d1>100 | y1 | 1 | 0 |
| 2017-01-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 62 |
| 2017-02-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 79 |
| 2017-03-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 94 |
| 2017-04-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 105 |
| 2017-05-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 128 |
| 2017-06-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 134 |
| 2017-07-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 113 |
| 2017-08-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 109 |
| 2017-09-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 83 |
| 2017-10-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 75 |
| 2017-11-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 68 |
| 2017-12-01 | Zch_Stampfenbachstrasse | O3_98%_min30 | m1 | µg/m3 | 72 |
_inputs_
Der Parameter _inputs_ ist ein Hilfskonstrukt wie
default_statistic. Ein Problem bei mehrstufigen
Berechnungen ist, dass unter Umständen bereits Resultate im Input für
weitere Berechnungen enthalten sind. In folgenden Bespiel wird zuerst
aus den 30min Werten d1 Mittelwerte, Anzahl Datenpunkte, min und max
berechnet für d1, m1 und y1. Danach sollte das Tagesmaximum für das Jahr
bestimmt werden:
# Ein Parameter reicht für die Veranschaulichung
O3 <- dplyr::filter(data, .data$parameter == "O3")
statstable <- tibble::tribble(
~parameter, ~statistic, ~from, ~to,
"default_statistic", "mean, n, min, max", "input", "d1, m1, y1",
"default_statistic", "max", "d1", "y1"
)
# Keine Berücksichtung der Datenverfügbarkeit
stats <- calculate_statstable(O3, statstable, data_thresh = 0, max_gap = NULL)
stats$y1
#> # A tibble: 8 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3 y1 µg/m3 4.53e+1
#> 2 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_min30 y1 1 1.75e+4
#> 3 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_min_min… y1 µg/m3 2.44e-1
#> 4 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_min… y1 µg/m3 1.54e+2
#> 5 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_d1 y1 µg/m3 1.16e+2
#> 6 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_min3… y1 1 4.8 e+1
#> 7 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_min_min… y1 µg/m3 9.00e+1
#> 8 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_min… y1 µg/m3 1.54e+2Wird für die Berechnung des maximalen Tagesmittelwert des Jahres
_inputs_ statt default_statistic verwendet,
werden nur die Statistiken für Parameter berechnet die in den Input
Daten enthalten sind.
statstable <- tibble::tribble(
~parameter, ~statistic, ~from, ~to,
"default_statistic", "mean, n, min, max", "input", "d1, m1, y1",
"_inputs_", "max", "d1", "y1"
)
# Keine Berücksichtung der Datenverfügbarkeit
stats <- calculate_statstable(O3, statstable, data_thresh = 0, max_gap = NULL)
stats$y1
#> # A tibble: 5 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3 y1 µg/m3 4.53e+1
#> 2 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_nb_min30 y1 1 1.75e+4
#> 3 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_min_min… y1 µg/m3 2.44e-1
#> 4 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_min… y1 µg/m3 1.54e+2
#> 5 2017-01-01 00:00:00 Zch_Stampfenbachstrasse O3_max_d1 y1 µg/m3 1.16e+2Im Allgemeinen dürfte die explizite Definition der Statistiken für
jeden Parameter gegenüber der Verwendung von
default_statistic und _inputs_ vorzuziehen.
Ansonsten kann das Resultat unerwartete Ergebnisse enthalten. Die
Definition der statstable kann auch in einer Textdatei oder Exceldatei
erfolgen und dann eingelesen werden, statt im Code definiert werden.
An dieser Stelle noch die Berechnungen aller Statistiken im aqmet Store. Aus 21 Input Grössen werden 419 Statistiken berechnet:
| parameter | statistic | from | to |
|---|---|---|---|
| CO, NO, NOx, NO2, O3, SO2 | mean | input | h1,d1,m1,y1 |
| CO, NO, NOx, NO2, O3, SO2 | max, min, n | input | d1,m1,y1 |
| CO, NO, NOx, NO2, SO2 | perc95 | input | m1,y1 |
| O3 | perc02, perc98 | input | m1,y1 |
| O3 | max, min, n, n>120, n>160, n>180, n>200, n>240 | h1 | d1,m1,y1 |
| NO2 | max, n, n>200 | h1 | d1,m1,y1 |
| O3 | mean | h1 | h8gl |
| CO | mean | h1 | h8gl |
| CO | max | h8gl | y1 |
| O3 | max, n>120, n>160, n>180, n>200, n>240 | h8gl | y1 |
| CO, NO, NOx, NO2, O3, SO2 | min, max, n | d1 | m1,y1 |
| CO | n>8 | d1 | m1,y1 |
| SO2 | n>100 | d1 | m1,y1 |
| NO2 | n>80 | d1 | m1,y1 |
| O3 | n>120, n>160, n>200, n>180, n>240, n>65 | d1 | m1,y1 |
| O3_max_h1 | n>120, n>160, n>180, n>200, n>240 | d1 | m1,y1 |
| O3_98%_min30 | max, n>100 | m1 | y1 |
| EC1.0, EC2.5, PM10, PM2.5, PN | mean | input | h1,d1,m1,y1 |
| EC1.0, EC2.5, PM10, PM2.5, PN | max, min, n | input | d1,m1,y1 |
| EC1.0, EC2.5, PM10, PM2.5, PN | perc95 | input | m1,y1 |
| EC1.0, EC2.5, PM10, PM2.5, PN | min, max, n | d1 | m1,y1 |
| PM10 | n>50 | d1 | m1,y1 |
| PM2.5 | n>25 | d1 | m1,y1 |
| WD | wind.direction | input | h1,d1,m1,y1 |
| WVs | wind.speed_scalar | input | h1,d1,m1,y1 |
| WVv | wind.speed_vector | input | h1,d1,m1,y1 |
| WD | n | input | d1,m1,y1 |
| WVs | max, min, n | input | d1,m1,y1 |
| WVv | max, min, n | input | d1,m1,y1 |
| Hr, p, StrGlo, T | mean | input | h1,d1,m1,y1 |
| Hr, p | min,max | input | d1 |
| T | min, max, n | input | d1,m1,y1 |
| T | min, max | h1 | d1,m1,y1 |
| T | min, max, n | d1 | m1,y1 |
| StrGlo | max, n | input | d1,m1,y1 |
| Hr, p | n | input | d1,m1,y1 |
| RainDur, RainSum, SunDur | sum | input | h1,d1,m1,y1 |
| RainDur, RainSum, SunDur | n | input | d1,m1,y1 |
| RainDur, RainSum, SunDur | max | d1 | y1 |
Umwandlung von Volumen- und Massenkonzentrationen
Die Umwandlung der Konzentrationen erfolgt äquivalent zu den
Berechnungen in der AIRMO. Sollen andere Konstanten verwendet werden,
ist die Dokumentation von convert_set_R() zu konsultieren.
Die Funktionen convert_conc() und
convert_conc_multiple() verfügen beide über Varianten wie
die umgewandelten Daten zurückgegeben werden. Definiert wird dies mit
dem Argument method. Bei “return” werden nur die umgewandelten Daten
zurückgegeben, bei “append” werden sie am Ende angehängt und zuletzt bei
“replace” werden die ursprünglichen Daten ersetzt.
data <- read_airmo_csv(fs::path(examples_path, "Zch_Stampfenbachstrasse_min30_2013_Jan.csv"))
# Behalte nur Volumenkonzentrationen
data <- dplyr::filter(data, .data$unit == "ppb" | .data$unit == "ppm")
convert_conc(data, "NO", "ppb", "µg/m3", method = "return")
#> # A tibble: 1,484 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO min30 µg/m3 79.3
#> 2 2013-01-01 00:30:00 Zch_Stampfenbachstrasse NO min30 µg/m3 81.8
#> 3 2013-01-01 01:00:00 Zch_Stampfenbachstrasse NO min30 µg/m3 75.6
#> 4 2013-01-01 01:30:00 Zch_Stampfenbachstrasse NO min30 µg/m3 81.2
#> 5 2013-01-01 02:00:00 Zch_Stampfenbachstrasse NO min30 µg/m3 101.
#> 6 2013-01-01 02:30:00 Zch_Stampfenbachstrasse NO min30 µg/m3 61.4
#> 7 2013-01-01 03:00:00 Zch_Stampfenbachstrasse NO min30 µg/m3 63.6
#> 8 2013-01-01 03:30:00 Zch_Stampfenbachstrasse NO min30 µg/m3 61.0
#> 9 2013-01-01 04:00:00 Zch_Stampfenbachstrasse NO min30 µg/m3 63.6
#> 10 2013-01-01 04:30:00 Zch_Stampfenbachstrasse NO min30 µg/m3 70.7
#> # ℹ 1,474 more rows
conversions <- tibble::tribble(
~parameter, ~from, ~to,
"CO", "ppm", "mg/m3",
"NO", "ppb", "µg/m3",
"O3", "ppb", "µg/m3",
"NO2", "ppb", "µg/m3",
"SO2", "ppb", "µg/m3"
)
convert_conc_multiple(data, conversions, method = "return") %>%
dplyr::arrange(.data$starttime)
#> # A tibble: 7,424 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.782
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO min30 µg/m3 79.3
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 min30 µg/m3 3.48
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO2 min30 µg/m3 63.0
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse SO2 min30 µg/m3 11.7
#> 6 2013-01-01 00:30:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.828
#> 7 2013-01-01 00:30:00 Zch_Stampfenbachstrasse NO min30 µg/m3 81.8
#> 8 2013-01-01 00:30:00 Zch_Stampfenbachstrasse O3 min30 µg/m3 3.40
#> 9 2013-01-01 00:30:00 Zch_Stampfenbachstrasse NO2 min30 µg/m3 64.7
#> 10 2013-01-01 00:30:00 Zch_Stampfenbachstrasse SO2 min30 µg/m3 12.2
#> # ℹ 7,414 more rows
convert_conc_multiple(data, conversions, method = "append") %>%
dplyr::arrange(.data$starttime)
#> # A tibble: 16,332 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO min30 ppm 0.672
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO min30 ppb 63.6
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO2 min30 ppb 33.0
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NOx min30 ppb 96.5
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 min30 ppb 1.75
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse SO2 min30 ppb 4.41
#> 7 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.782
#> 8 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO min30 µg/m3 79.3
#> 9 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 min30 µg/m3 3.48
#> 10 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO2 min30 µg/m3 63.0
#> # ℹ 16,322 more rows
convert_conc_multiple(data, conversions, method = "replace") %>%
dplyr::arrange(.data$starttime)
#> # A tibble: 8,908 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NOx min30 ppb 96.5
#> 2 2013-01-01 00:00:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.782
#> 3 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO min30 µg/m3 79.3
#> 4 2013-01-01 00:00:00 Zch_Stampfenbachstrasse O3 min30 µg/m3 3.48
#> 5 2013-01-01 00:00:00 Zch_Stampfenbachstrasse NO2 min30 µg/m3 63.0
#> 6 2013-01-01 00:00:00 Zch_Stampfenbachstrasse SO2 min30 µg/m3 11.7
#> 7 2013-01-01 00:30:00 Zch_Stampfenbachstrasse NOx min30 ppb 99.4
#> 8 2013-01-01 00:30:00 Zch_Stampfenbachstrasse CO min30 mg/m3 0.828
#> 9 2013-01-01 00:30:00 Zch_Stampfenbachstrasse NO min30 µg/m3 81.8
#> 10 2013-01-01 00:30:00 Zch_Stampfenbachstrasse O3 min30 µg/m3 3.40
#> # ℹ 8,898 more rowsDie Funktion calculate_mass_concentrations() ist eine
Hilfsfunktion, die aus Volumenkonzentrationen für CO, NO, NO2, NO2, SO2
in einem tibble automatisch die Massenkonzentrationen berechnet. Mit dem
Argument keep_ppb können optional die Volumenkonzentration behalten
werden. Das Default vorgehen ist die Volumenkonzentrationen zu
ersetzen.
Pluck
Die Pluck Funktionen sind Wrapper um dplyr::filter().
Sie ermöglichen dem Nutzer eine komfortable Filterung von Daten im rolf
Format. Als Beispiel nehmen wir die Daten aus dem mehrfach Meteoschweiz
Export, dieser enthaltet eine Vielzahl von verschiedenen Stationen,
Intervalle und Parameter:
fn <- system.file("extdata", "smn_multi.txt", package = "rOstluft.data")
data <- read_smn_multiple(fn) %>%
dplyr::arrange(starttime)
pluck_parameter(data, "ta1towb0")
#> # A tibble: 6 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2010-01-01 00:00:00 UEB ta1towb0 h1 NA 3.7
#> 2 2010-01-01 01:00:00 UEB ta1towb0 h1 NA 3.5
#> 3 2010-01-01 02:00:00 UEB ta1towb0 h1 NA 3.2
#> 4 2010-01-01 03:00:00 UEB ta1towb0 h1 NA 3.1
#> 5 2010-01-01 04:00:00 UEB ta1towb0 h1 NA 3.3
#> 6 2010-01-01 05:00:00 UEB ta1towb0 h1 NA 3.3
# entpackt automatisch Vectors
pluck_site(data, c("KLO", "UEB")) %>% dplyr::slice(41:46)
#> # A tibble: 6 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2010-01-01 05:00:00 UEB fk1towb0 h1 NA 1.9
#> 2 2010-01-01 05:00:00 UEB ta1towb0 h1 NA 3.3
#> 3 2018-01-01 00:00:00 UEB uretowhs h1 NA 85.1
#> 4 2018-01-01 00:50:00 KLO dkl010z0 min10 NA 224
#> 5 2018-01-01 00:50:00 KLO fkl010z0 min10 NA 5.7
#> 6 2018-01-01 00:50:00 KLO fkl010z1 min10 NA 9.6
# supported splicing mit den rlang operator !!!
intervals = c("h1", "d1")
pluck_interval(data, !!!intervals) %>% dplyr::slice(40:45)
#> # A tibble: 6 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2010-01-01 05:00:00 UEB dk1towb0 h1 NA 129
#> 2 2010-01-01 05:00:00 UEB fk1towb0 h1 NA 1.9
#> 3 2010-01-01 05:00:00 UEB ta1towb0 h1 NA 3.3
#> 4 2017-12-31 01:00:00 RHW wkcap1d0 d1 NA 2
#> 5 2017-12-31 01:00:00 RHW wkcap2d0 d1 NA 11
#> 6 2017-12-31 01:00:00 RHW wkcap3d0 d1 NA 21
# pluck_year mit einer sequence
pluck_year(data, 2010:2018) %>% dplyr::slice(51:56)
#> # A tibble: 6 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2017-12-31 01:00:00 RHW wkwtp1d0 d1 NA 1
#> 2 2017-12-31 01:00:00 RHW wkwtp2d0 d1 NA 9
#> 3 2017-12-31 01:00:00 RHW wkwtp3d0 d1 NA 9
#> 4 2018-01-01 00:00:00 UEB uretowhs h1 NA 85.1
#> 5 2018-01-01 00:50:00 ARH dkl010z0 min10 NA 168
#> 6 2018-01-01 00:50:00 ARH fkl010z0 min10 NA 1.1
# NA Werte werden gefiltert
pluck_unit(data, "hPa")
#> # A tibble: 6 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2019-01-31 00:50:00 TAE prestas0 min10 hPa 936.
#> 2 2019-01-31 01:00:00 TAE prestas0 min10 hPa 936
#> 3 2019-01-31 01:10:00 TAE prestas0 min10 hPa 936.
#> 4 2019-01-31 01:20:00 TAE prestas0 min10 hPa 936.
#> 5 2019-01-31 01:30:00 TAE prestas0 min10 hPa 936.
#> 6 2019-01-31 01:40:00 TAE prestas0 min10 hPa 936.
# Verkettbar mittels pipes
data %>%
pluck_site("KLO", "UEB") %>%
pluck_parameter("gre000z0") %>%
pluck_year(2010:2018) %>%
dplyr::slice(1:6)
#> # A tibble: 6 × 6
#> starttime site parameter interval unit value
#> <dttm> <fct> <fct> <fct> <fct> <dbl>
#> 1 2018-01-01 00:50:00 KLO gre000z0 min10 NA 1
#> 2 2018-01-01 00:50:00 UEB gre000z0 min10 NA 3
#> 3 2018-01-01 01:00:00 KLO gre000z0 min10 NA 1
#> 4 2018-01-01 01:00:00 UEB gre000z0 min10 NA 3
#> 5 2018-01-01 01:10:00 KLO gre000z0 min10 NA 1
#> 6 2018-01-01 01:10:00 UEB gre000z0 min10 NA 2Koordinatentransformationen
Für die Darstellung von Elementen mit Koordinaten im Bezugsystem LV95 oder LV03 auf Karten ist es oft notwendig
diese nach WSG84 zu transformieren.
Die Funktionen rOstluft::transform_LV95_to_WSG84() und
rOstluft::transform_WSG84_to_LV95() sind Wrapper um die
allgemeine Funktion rOstluft::transform_crs().
meteoschweiz <- readRDS(fs::path(examples_path, "meta_smn.rds"))
meteoschweiz <- dplyr::distinct(meteoschweiz, site, x, y)
transform_LV95_to_WSG84(meteoschweiz) %>% head
#> # A tibble: 6 × 5
#> site x y lon lat
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 Aadorf/Tänikon 2710517 1259824 8.90 47.5
#> 2 Altenrhein 2760382 1261386 9.57 47.5
#> 3 BadRagaz 2756910 1209350 9.50 47.0
#> 4 Chur 2759465 1193152 9.53 46.9
#> 5 Ebnat-Kappel 2726347 1237176 9.11 47.3
#> 6 Glarus 2723755 1210567 9.07 47.0
transform_crs(meteoschweiz, c(lon = "x", lat = "y"),
sp::CRS("+init=epsg:2056"), sp::CRS("+init=epsg:4326"),
append = FALSE) %>% head
#> Warning in CPL_crs_from_input(x): GDAL Message 1: +init=epsg:XXXX syntax is
#> deprecated. It might return a CRS with a non-EPSG compliant axis order.
#> # A tibble: 6 × 3
#> site lon lat
#> <chr> <dbl> <dbl>
#> 1 Aadorf/Tänikon 8.90 47.5
#> 2 Altenrhein 9.57 47.5
#> 3 BadRagaz 9.50 47.0
#> 4 Chur 9.53 46.9
#> 5 Ebnat-Kappel 9.11 47.3
#> 6 Glarus 9.07 47.0NOAA Hysplit Trajektorien
Neben dem Ostluft AWS Store aqmet existiert noch ein Store mit hysplit Trajektorien von 1980 bis zum jeweiligen Vormonat des aktuellen Datums für die Standorte Zürich Kaserne und St. Gallen Blumenbergplatz. Die Trajektorien sind jeweils für 01:00 und 13:00 UTC+1 (=Luftmassen Herkunft) gerechnet.
Beim Format hysplit wird für die Abfrage nur die site
und das year benötigt:
hysplit_store <- storage_s3_rds("hysplit_tutorial", format_hysplit(), "rostluft", "hysplit")
#> store hysplit_tutorial initialized under 'C:\Users\tom\AppData\Local/rOstluft/hysplit_tutorial'
# nutze den vordefinierten store (name ist optional, default ist "hysplit")
hysplit_store <- store_hysplit("hysplit_tutorial")
# hole die Trakektorien für Zürich Kaserne von 2018
traj <- hysplit_store$get(site = "ZH_Kaserne_hysplit", year = 2018)
# plotte die Daten mit openair
openair::trajPlot(openair::selectByDate(traj, start = "2018-01-01", end = "2018-01-05"))
Lokale Daten löschen
Werden die Daten lokal nicht mehr benötigt, kann mit der Funktion
$destroy() des Store Objektes die Daten gelöscht werden. Zu
beachten ist, dass der AWS S3 Store als read only initialisiert wurde.
Um ihn löschen zu können muss dies geändert werden. Um ein
versehentliches löschen durch Autocomplete zu verhindern, muss der
String “DELETE” als Argument übergeben werden
store$read.only = FALSE
store$destroy("DELETE")
#> Cache for Store tutorial destroyed
my_store$destroy("DELETE")
#> Store eigene_daten destroyed
hysplit_store$read.only = FALSE
hysplit_store$destroy("DELETE")
#> Cache for Store hysplit_tutorial destroyed